Com a proposta de processar dados de transcriptoma para o estudo das funções e interações das proteínas nas células, a Bioinformática impulsiona descobertas ímpares no mundo biológico.
A Bioinformática e a Biologia Sistêmica (que pretende entender os organismos biológicos em todos os seus níveis) têm, entre seus desafios, a inferência de redes de regulação gênica, destinada a deduzir as interações existentes entre os genes/proteínas por meio de dados de expressão gênica, que mede o quanto um gene pode estar sendo traduzido em proteína.
Na Faculdade de Computação (FACOM) da UFMS, alguns docentes e pós-graduandos debruçam-se sobre estudos na área. O professor Carlos Henrique Aguena Higa comanda a pesquisa “Inferência de redes de regulação gênica a partir de dados biológicos” e irá expor, no próximo mês de julho, dois trabalhos da área na 18th International Conference on Computational Science and Its Applications, em Melbourne, Austrália.
“Para o organismo funcionar bem a célula precisa estar em equilíbrio. Existe um processo de regulação dos elementos químicos que fazem parte da célula, como as proteínas, e isso não acontece de maneira isolada. Essas proteínas acabam interagindo entre si para realizar uma função dentro da célula. O foco da pesquisa é exatamente gerar hipóteses de regulação gênica, para auxiliar outros pesquisadores, um bioquímico por exemplo, a descobrir quais proteínas interagem com quais”, explica o professor.
Traduzindo para a Computação, a partir de um grafo em que os vértices (genes) são conhecidos, é preciso inferir as arestas direcionadas, ou seja, as regulações gênicas. “É como se cada gene/proteína fosse um ponto e é preciso conectar esses pontos através de retas, que chamamos de arestas, e o que temos de informação são os dados de expressão gênica. Por isso nomeamos esse processo de inferência de redes de regulação gênica”, expõe Carlos Higa.
Matemática x biologia
Para realizar essas inferências, são usados modelos matemáticos, como as redes Booleanas, redes Bayesianas, equações diferenciais, em conjunto com diversas metodologias e algoritmos. “Nos anos 90 surgiu a tecnologia de microarrays e hoje temos as tecnologias de sequenciamento de nova geração. Conseguimos obter dados de expressão gênica de maneira mais rápida, barata e confiável, e a partir daí aplicar metodologias para a inferência das redes de regulação gênica”, diz o professor.
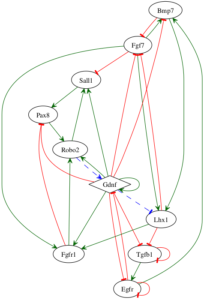
Inicialmente são feitos testes com dados sintéticos, artificiais, para testar a metodologia. Depois, é aplicada em dados reais. Na Facom, o processo foi realizado em experimentos com dados do ciclo celular da levedura (Saccharomyces cervisiae) e do desenvolvimento do rim humano. Um trabalho futuro é estabelecer uma colaboração com Embrapa para aplicar a metodologia em dados do capim Panicum maximum sob estresse hídrico, de forma a saber como a planta se comporta quando falta água.
O propósito final é saber se a partir desse algoritmo, dessa metodologia, consegue-se formular hipóteses de regulação gênica, que depois podem ser testadas em bancada.
“Os problemas são apresentados por biólogos ou bioquímicos, que possuem dados porém encontram dificuldades em processá-los. Assim, procuram pesquisadores da Bioinformática, que os processa, e retorna os resultados, mostrando quais são as redes inferidas. O profissional da área biológica precisa então analisar e verificar o que faz sentido ou não. É isso o que faz essa pesquisa de inferência de redes”, afirma o professor.
Agora, em uma nova etapa, o professor pretende desenvolver novas pesquisas para o controle de redes de regulação gênica, com o propósito de apontar o que é preciso mudar na rede para que o organismo passe a ter outro comportamento.
“Após a inferência da rede de regulação gênica de uma doença, por exemplo, o bioquímico verifica aquela interação e nos questiona o que é preciso mudar nessa rede para a célula passar a se comportar de outro jeito, sair desse estado, parar de produzir a proteína que está causando o dano. Essa é um outro campo de pesquisa, conhecido como controle de redes de regulação gênica”, explica o professor.
Proteína é o produto gênico final do genoma. O que diferencia uma célula de outra em um mesmo organismo são as proteínas que estão sendo produzidas nas células e suas interações. Quais as interações presentes que é a grande questão a ser respondida.
Interesse na área
É crescente o interesse dos alunos pela Bioinformática, segundo Carlos Higa. “Está cada vez mais atraente. Conforme a tecnologia vai se desenvolvendo você consegue gerar mais dados de maneira mais rápida e barata. Antigamente, para sequenciar o DNA, por exemplo, lavava-se muito tempo. A primeira vez em que o genoma humano foi sequenciado levou-se anos, a um custo muito alto. Hoje, é possível sequenciar genomas em horas, minutos, a um custo muito mais baixo”, completa o professor.
A Bioinformática demanda de seus entusiastas interesse em conhecimento multidisciplinar, passando pela Computação, Matemática, Biologia, Química e outras áreas relacionadas. Segundo o professor, existem diversos cursos de pós-graduação em Bioinformática no país. “Na Computação, modelamos os problemas de forma matemática, com estruturas matemáticas, como por exemplo, grafos. Mas na Biologia não é bem assim pois não é uma ciência exata. Por isso é preciso ter uma visão multidisciplinar e colaboração com pesquisadores de outras áreas”, diz.
Paula Pimenta


